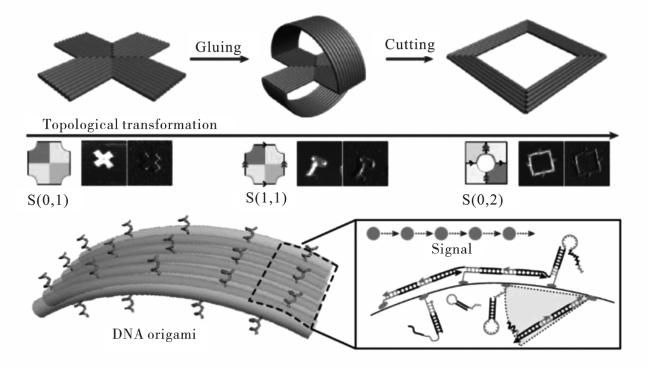
1 DNA电路
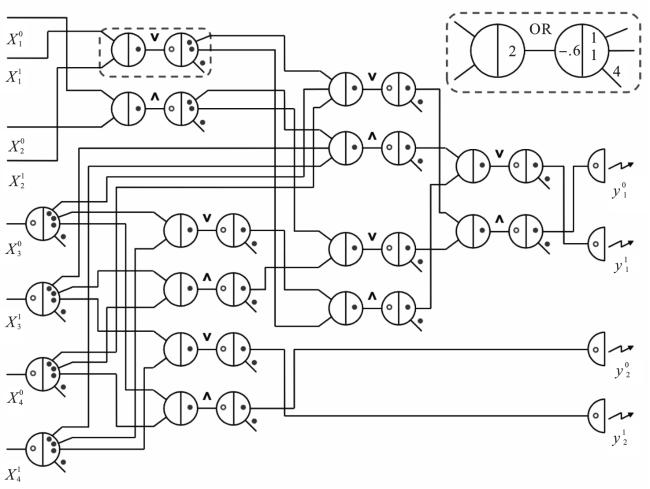
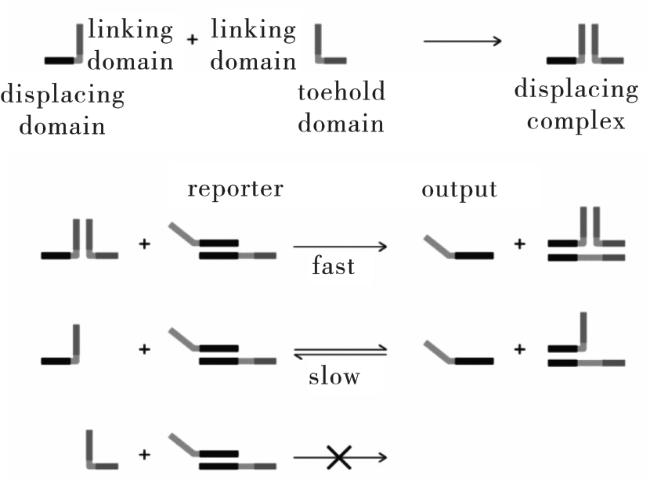
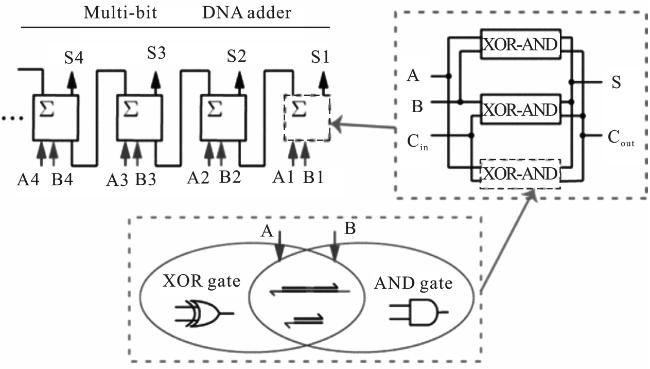
1.1 基于DNA链置换的DNA电路
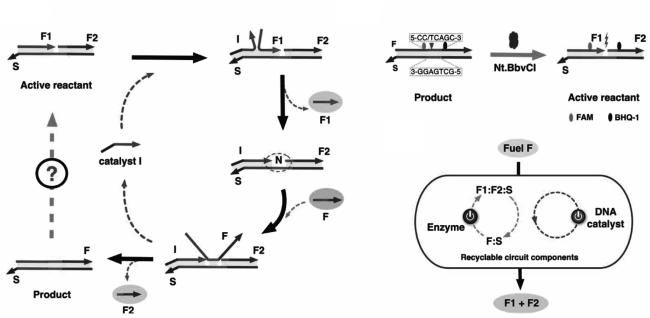
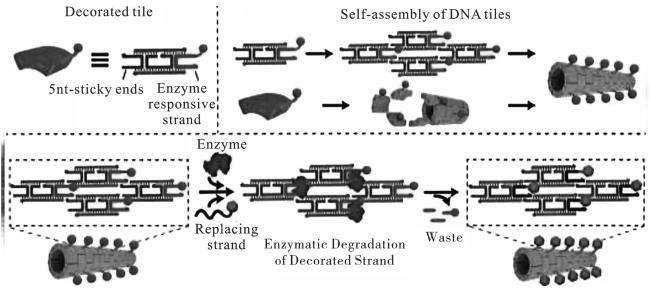
1.2 蛋白质酶介导的DNA电路
表1 应用于DNA电路的常见蛋白质酶 |
| 类型 | 概念 | 基本特性 | 用途 |
|---|---|---|---|
| DNA聚合酶 | 在DNA或RNA模版指导下,以4种脱氧核糖核苷酸为底物,在引物3' —羟基末端聚合DNA链的酶 | 需模版,需引物,具有5'→3' 聚合酶活性,具有5'⇌3'外切酶 活性 | 缺口平移法制备DNA分子杂交探针,DNA序列测定 |
| DNA连接酶 | 能够催化DNA中3' —羟基和5' —磷酸基末端之间形成3',5' —磷酸二酯键的酶 | 可连接带匹配黏性末端的DNA分子,也可使平端的双链DNA分子相互连接 | 连接2个DNA分子或片段 |
| 限制性内切酶 | 能识别双链DNA分子中特定核苷酸序列,并在识别序列内或附近特异切割双链DNA的酶 | 识别序列具回文结构,切割末端有黏端和平端 | 识别DNA特定序列,切割DNA分子 |